- 2023-07-11 16:26:21 来源:贝克曼库尔特商贸(中国)有限公司 浏览量:274次
- 【导读】合成生物学的研究内容主要是采用工程化方法和工程化开发平台,用生物分子及由生物分子构成的不同层次的生物组件,设计、创造出适应不同目的的生物装置和系统。
-
合成生物学的研究内容主要是采用工程化方法和工程化开发平台,用生物分子及由生物分子构成的不同层次的生物组件,设计、创造出适应不同目的的生物装置和系统。其主要原则是设计、评估、以及遗传部分的标准化。一个挑战是部分设计即修改遗传序列作为不同的单位,最 终结合成更复杂的遗传回路,在目标生物体中产生所需的功能。在合成生物学应用中,通过设计-构建-测试-学习(DBTL)循环对遗传部分或生物体连续通过迭代过程进行改进。本次小贝给大家总结了几篇合成生物学自动化方案,这些方案为我们介绍了如何通过计算机辅助设计软件设计(D)新的DNA序列以获得所需的特性,物理构建(B)新的构造变体,以及在实验室中使用分析仪器进行测试(T),计算机建模用于学习(L)和预测遗传部分特征。
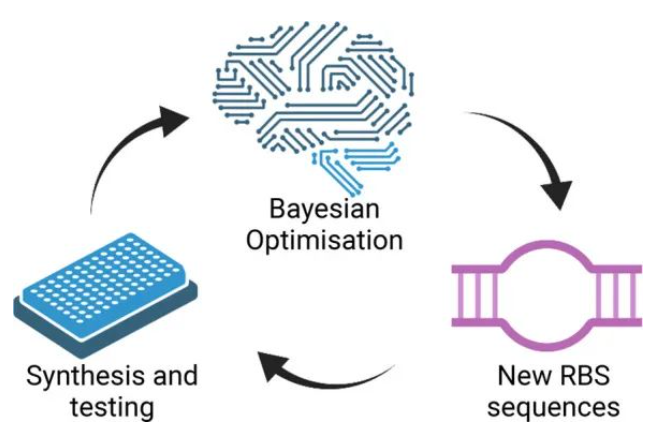
01、机器学习引导细菌核糖体结合位点的批量设计
优化基因表达水平是生物体设计过程重要的部分,可通过工程化转录和翻译控制元件实现对这一过程的精细控制。大多数计算机模型基于相关分子(DNA、RNA、蛋白质等)的热力学特性或经验得出的描述相关设计特性的值,如细菌中控制蛋白表达的关键遗传元件——核糖体结合位点(RBS)的翻译起始率(TIR)。然而,当前由于缺乏可靠的设计方法,小遗传元件的从头设计由于顺序和性能之间的未知关系而具有挑战性。为了解决这个问题,Mengyan Zhang等人创建了一套机器学习指导DBTL循环用于细菌RBS的实验设计以证明如何使用相对小的、高质量的数据集相对可靠地设计较小的遗传部件。值得注意的是,作者还将这些机器学习算法与实验室自动化和高通量流程集成在一起,实现可靠的数据生成,如使用贝克曼库尔特Echo纳升级移液工作站将1 μL反向引物加入到96孔板中构建PCR反应体系,以及在后续细胞培养分析中,将1 μL浓度为0.1M的高效诱导剂IPTG转移到96孔细胞培养板中,从而提高移液精确度降低误差,提高分析准确度;随后通过在四个DBTL周期中测试总共450个RBS变体,验证了RBS的翻译起始率高达34%,这一结果等于或超过基准RBS。表明机器学习是一种强大的工具能够用于设计RBS,也为更复杂的遗传元件铺平了道路。
02、basicsynbio和BASIC SEVA集合:用于建立DNA组装方法的软件和载体
DNA组装是合成生物学和生命科学DBTL循环必不可少的工具。虽然当前有大量的DNA组装方法可供研究人员选择,但如何选择合适的方法则取决于各种因素,例如对于包含禁用的限制性位点的自由度,组件库的可用性或对高精度的需求。实现高通量和分层组装理想的情况是建立标准化和模块化的DNA组装方法,在高精度、经济高效地生成大量结构的同时还能够促进不同设计间部件的重复使用。
幂等克隆生物部件组装标准(BASIC) DNA组装是一种标准化的DNA组装方法,它利用模块化组件和接头作为功能单元。该方法受益于几个理想的属性,包括单组件存储格式以及每轮组装多达 14 个部件和接头,同时精度 >90%;可以免费用于学术和商业用途,而且易于自动化工作流,并执行分层程序集。接头可以编码诸如核糖体结合位点(RBS)和融合蛋白接头之类的功能序列,因此在单轮组装中可以构建不同的结构。BASIC与基于Golden Gate组装的模块化方法相比具有优势,它使用多种限制性内切酶,并且不支持不符合标准转录单元的组件,例如操纵子。BASIC DNA组装已成功应用于合成生物学和生命科学研究的多个领域,包括组合途径工程、合成操纵子和小的非编码RNA回路设计,RBS优化、融合蛋白工程和多重引导RNA表达等方向。
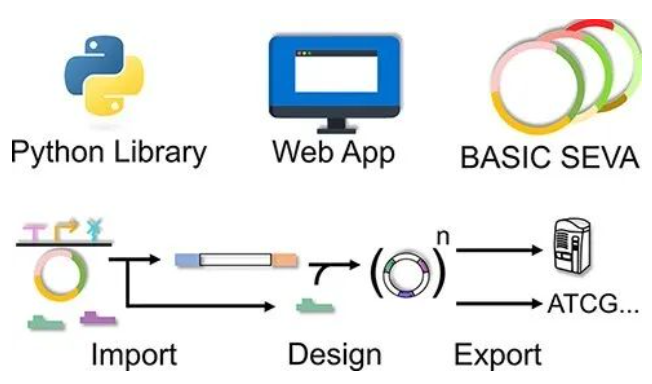
Matthew C Haines等发布了包含Web应用程序和Python包的basicsynbio开源软件,可通过简单的拖放操作或编程方式实现BASIC构造设计。用户可以在设计新部件和组件时访问常用的BASIC组件和接头,并对常见错误进行异常处理;同时为下一阶段的验证导出序列数据并为手动或贝克曼库尔特生命科学Echo纳升级移液平台移液创建指令。如clip反应和MagBead纯化步骤完成后,将纯化的clip反应产物转移到符合Echo®标准的384孔聚丙烯微孔板中,也可以通过在Echo上执行脚本来将Echo®聚丙烯微6孔板上的ddH2O和10×的组装缓冲溶液通过声波移液与纯化的clip反应产物混合。随后完成组装,转化,并接种在含有抗生素的LB琼脂上,以及挑选粉红色菌落等步骤。
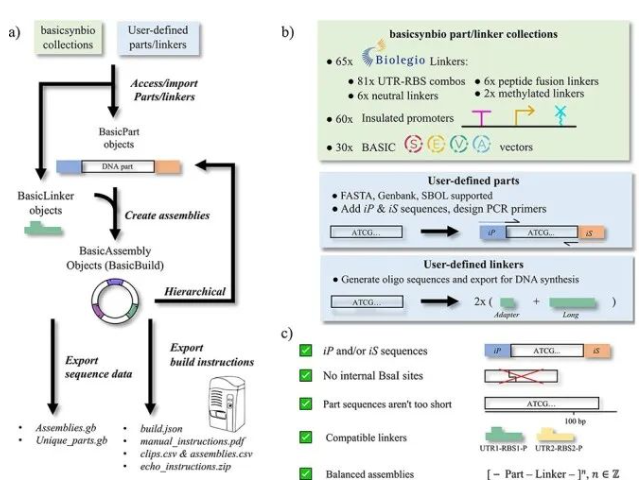
为了证明灵活性,研究人员演示Basicsynbio使用序列组装包含六种抗生素耐药性标志物之一和来自不同相容组的五种复制来源的30个载体(包括来自标准欧洲载体架构(SEVA)的模块)。作者编写了将 BasicBuild 类对象解析为.pdf格式的手动指令和贝克曼库尔特生命科学Echo 液体处理平台指令的函数,通过计算所有组件所需的三个参数指导BASIC工作流程的clip反应设置和装配阶段的液体处理操作,使clip反应体系的部分组分浓度为2.5nM,从而使效率实现最 大化。

使用basicsynbio Python软件包和Web App,用户可以访问常用的部件和接头,在导出序列数据和构建指令的同时稳健地设计新的部件,接头和组件。结合其他可接近的部件和接头,用户可以轻松可靠地设计大量组件,为设计生物工程应用的组件提供了强大的环境,从而实现在合成生物学和生命科学领域的应用。
03、高通量、经济高效的结构DNA组装验证
无论是基因合成(∼1 kb),途径工程(∼10 kb)还是合成基因组(>100 kb),“搭积木式”的DNA“组装”仍然是合成生物学的基石。尽管用于DNA组装的技术取得了许多进步,但随着规模的扩大,作为一项基本任务以确保基因型与表型准确映射的DNA构建体验证的成本也越来越高昂,并且面临着逻辑方面的挑战。Yandi Dharmadi等人每月设计和制造数百到数千个独特的构建块和组件,结合反应的多样性(构建体数量×克隆数量×连接数量),通过测序或junction-PCR验证数千个DNA构建体成本就会变得高昂和/或逻辑上难以处理,一种廉价、快速和可靠的验证方法是绝 对必要的。为此,Yandi Dharmadi等人开发出一种限制性酶切实现结构性DNA组装验证的全面、高通量解决方案,这一方案使用详尽的计算机酶筛选、质粒DNA的滚动循环扩增、毛细管电泳和自动消化模式识别来实现。实验流程设计上,384孔格式在贝克曼库尔特生命科学Biomek FXP自动化工作站上完成程序化的液体转移(2-200μl),使用Qpix 2高通量微生物克隆筛选系统完成大肠杆菌接种,采用低成本和稳健的96孔毛细管电泳方法进行验证,这一解决方案已成功用于筛选超过31,000个DNA构建体克隆,使每个样品综合成本<1美元,作为重要的一部分为建立工业规模DNA组装管线提供了非常大的帮助。
04、全E.coliTXTL工具箱3.0:无细胞合成生物学平台的新功能
点击标题可阅读原无细胞基因表达作为一种多用途生物工程技术,特点是快速的实验周转和更高的通量,以及高度的安全性,已被塑造成一种多功能且用户友好的工具,有助于调控元件和基因网络的快速原型设计,同时也广泛覆盖了不断增长的应用范围。随着DNA定向体外蛋白质合成范围的扩大,需要开发更强大的无细胞转录翻译(TXTL)平台来执行更大的DNA程序或提高无细胞生物制造能力。在David Garenne等人公布的一项工作中,一种专门为合成生物学开发的多用途无细胞表达系统——全大肠杆菌TXTL工具箱3.0提供了新的思路。
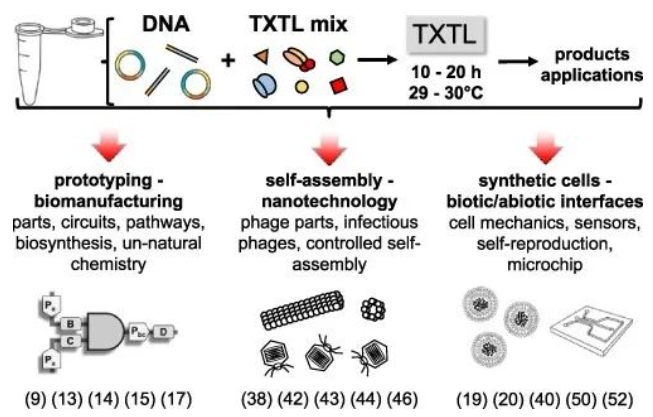
图 大肠杆菌TXTL系统及其应用概述
在该实验中,配置反应体系通过两种方式来实现,要么手工组装(10-20μl),要么使用贝克曼库尔特生命科学Echo纳升级移液工作站向96孔板上分液(2-5μl)以实现缩小反应体积,提高移液精确度。其次,无细胞反应除了补充60mM麦芽糊精之外,还补充了30mM d-核糖,并在29-30°C下进行无细胞反应。使用报告基因蛋白deGFP(d增强绿色荧光蛋白,在无细胞系统中更具可翻译性)(25.4 kDa,1 mg / ml = 39.37μM)孵育15-20小时后在酶标仪上测量间歇模式反应中产生的deGFP的终点荧光,标曲则使用带有His标签的纯重组eGFP,从而实现翻译效率的定量评估。在非补料分批模式反应中,deGFP的合成达到4mg / ml;在由负载有TXTL反应的脂质体组成的合成细胞中,当供给反应的化学构件通过膜通道扩散以促进与外部溶液的交换时,会产生浓度>8mg / ml的deGFP。说明这一TXTL系统能够通过提供独特的强度和特性来扩展当前的无细胞表达能力,对于测试调节元件和回路,生物制造生物制剂或构建合成细胞有非常大的帮助。
05、植物蛋白的生物铸造辅助表达和表征
合成生物学中的许多目标,包括生物合成途径的阐明和重构以及调节回路和网络工程,都需要蛋白质功能的知识。在植物中,大基因家族的普遍存在意味着将特定功能与单个蛋白质联系起来可能特别具有挑战性。目前蛋白质表征仍然是一个技术瓶颈,通常需要付出大量努力来优化表达和纯化方案。为了利用生物铸造厂的能力来加速DBTL周期,Quentin M Dudley等人提出了一种用于自动DNA组装和植物蛋白无细胞表达的工作流程,可加速优化并快速筛选酶活性。这包含开发一种与植物积块兼容的Golden Gate DNA组装工具箱,其中包含质粒受体,用于使用大肠杆菌或小麦胚芽裂解物进行无细胞表达,以及一组用于检测,纯化和改善表达/折叠的N端和C端标签部件;其次他们使用Echo纳升级液体处理平台优化了小型化无细胞反应的自动化组装,然后比较标签配置以提高标签表达。随后开发了一种基于荧光素酶的快速定量系统,该系统需要最 少的11个氨基酸标签,并演示了合成后标签的轻松去除。最 后,实验表明可以使用无细胞蛋白质合成反应进行几种功能测定,而无需蛋白质纯化。总之,DNA部件的自动组装和无细胞表达反应的结合显著提高实验通量,在测试和了解植物蛋白功能并使DNA部件在下游植物工程工作流程中直接重复使用提供了巨大帮助。
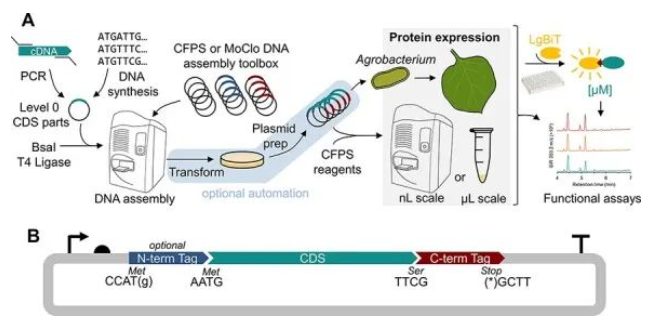
Echo用于植物蛋白的生物铸造辅助DNA组装和无细胞蛋白质合成(CFPS)的工作流程
使用Echo 550(贝克曼库尔特生命科学)通过两步策略配置2 μL无细胞蛋白质合成反应体系配置,第 一步将30-50 μL/质粒(或水)转移到符合Echo®标准的384孔聚丙烯源微孔板(384PP,P-05525)中。第二步,将已添加裂解物的无细胞蛋白合成试剂(CFPS)的预混液以65μL/孔转移至同一384PP板的空孔中。随后设置384PP_AQ_SP2 作为样品板类型使用Echo进行移液,定向转移到384 孔PCR目标板中,每孔1735 nL,每个来源孔可支持20-21个反应。然后,使用Echo® Cherry Pick软件(由.csv文件指导)设置384PP_AQ_BP2样品板类型将含有26.6 ng质粒(相当于最 终浓度为13.3 ng /μL)以及需要补的水共计265 nL分配到所有目标孔,随后目标板在室温下短暂离心,用粘性铝箔封膜,并在 PCR仪中30°C 孵育20小时(37°C热盖)。为了对比,在1.5 mL离心管中等比例放大CFPS反应。为了定量可溶性/总蛋白质,将反应物在4°C以21,000 g离心10分钟以沉淀不溶性蛋白质。
06、用于鉴定和表征代谢物诱导系统的全基因组方法
诱导基因表达系统是促进合成生物学发展的重要工具,作为基因编码生物传感器有望为诊断做出贡献,并彻底改变微生物细胞工厂开发领域。鉴于目前具有生物学意义的化合物的数量远远超过可用的生物传感器的数量,Erik K. R. Hanko等人通过开发一种通用的全基因组方法,来识别转录因子诱导基因表达系统来解决这一窘境。他们构建并验证了 15 种功能性生物传感器,提供了表征工作流程,展示了广泛的宿主适用性和在酶筛选中的实用性。
在这项研究的交叉反应筛选流程中,使用贝克曼库尔特生命科学自动化整合平台(整合了Biomek FXp自动化移液工作站(贝克曼库尔特生命科学)、SpectraMax 6i酶标仪(美谷分子)和Cytomat 2C振荡培养箱(Thermo))通过3块黑色微孔板来测试一种菌株对67种化合物(4个重复)的反应,自动化全流程实现相对诱导率的计算。使用整合系统软件SAMI EX(贝克曼库尔特)生成的工作流如下图所示:
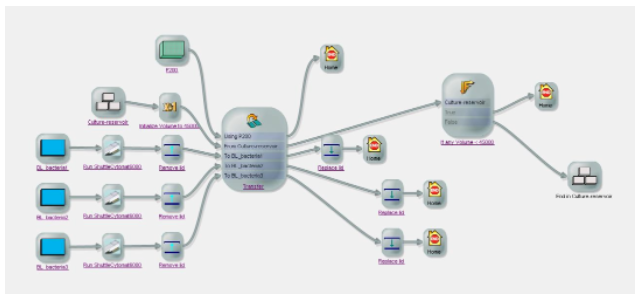
SEMI EX软件生成的工作流程图展示细菌培养自动化移液过程。
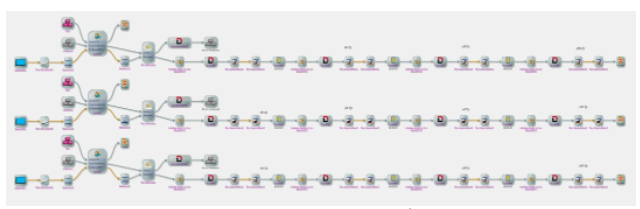
SEMI EX软件生成的工作流程图展示添加诱导因子,荧光强度测量和孵育培养过程。
通过向2 mL含氯霉素的基本培养基中接种单个新鲜转化的细菌细胞菌落来建立C. necator预培养。在30°C下200rpm振荡孵育18小时后,将细菌1:50稀释,在相同条件下再培养4小时直到OD600达到0.15-0.2。随后使用Biomek FXp自动化液体处理平台将 142.5 μL 细菌培养物分到黑色 96 孔微量滴定板中,并用 Biomek FXp将96孔深孔板中的10mM诱导剂转移7.5 μL到微量滴定板中的 C. necator 培养物中,将板转移至整合的Cytomat2振荡培养箱中,在30°C下600rpm诱导6、12和18小时,最 后使用整合的SpectraMax 6i酶标仪立即进行荧光和吸光度测量。
随着基因组数据的快速增长,这些创新为大幅增加生物可检测分子的数量提供了一个平台,对促进前瞻性工程工作有十分重要的意义。
07、酶工程:一种合成生物学方法,可实现更有效的文库生成和自动化高通量筛选
酶工程中有效的诱变策略往往依赖于小的、靶向的、高质量的突变体文库的生成。这种“智能”文库与筛选工作所施加的实际限制是一致的:虽然点突变文库很容易筛选,但仍需要创造性的解决方案来提高探索序列空间组合复杂程度的能力。Golden Gate基因组装策略使用的IIS型限制性内切酶的酶切位点在其识别序列之外,酶切后这些片段可以非常容易地无缝重组从而能够不受限制地设计独特的DNA片段。受这一策略的启发,Daniela Quaglia等人对南极念珠菌脂肪酶A(Cal-A)的三个不同区域对应的基因片段进行靶向或随机诱变,随后直接重组,实现了在一轮实验中组合不同诱变方法的产物,能够快速产生一系列不同的文库,从而促进定向进化,证明了其在设计生物催化剂方面的有利用途。
在贝克曼库尔特生命科学Biomek NXp自动化液体处理工作站的帮助下,将手动筛选南极念珠菌脂肪酶A(Cal-A)抗三丁酸甘油酯和橄榄油突变体转换为自动化,从而实现更高的通量。自动化流程使用倒好42 mL培养基的Nunc™ OmniTray™矩形培养皿进行涂布操作,培养皿堆叠在工作站台面上,通过脚本,从96孔板中逐列吸取20 μL饱和菌液,并通过轻轻接触琼脂表面来接种8 μL菌液,多余的菌液弃至乙醇Waste板中,而吸头随后可被重复用于接种相同的菌液。接种后的OmniTray板由机械臂抓手转移到耗材堆栈,并转移至30°C下孵育过夜。通过凝胶成像仪观察孵育的板并进行分析。这项研究通过通用的、自动化方法来视觉区分Cal-A表达E.coli BL21(DE3)对不同底物的水解活性,有助于提高对短链与长链脂肪酸底物选择性突变的筛选效率。
参考文献:
1、Zhang M, Holowko MB, Hayman Zumpe H, Ong CS. Machine Learning Guided Batched Design of a Bacterial Ribosome Binding Site. ACS Synth Biol. 2022 Jul 15;11(7):2314-2326. doi: 10.1021/acssynbio.2c00015. Epub 2022 Jun 15. PMID: 35704784; PMCID: PMC9295160.
2、Haines MC, Carling B, Marshall J, Shenshin VA, Baldwin GS, Freemont P, Storch M. basicsynbio and the BASIC SEVA collection: software and vectors for an established DNA assembly method. Synth Biol (Oxf). 2022 Oct 11;7(1):ysac023. doi: 10.1093/synbio/ysac023. PMID: 36381610; PMCID: PMC9664905.
3、Dharmadi Y, Patel K, Shapland E, Hollis D, Slaby T, Klinkner N, Dean J, Chandran SS. High-throughput, cost-effective verification of structural DNA assembly. Nucleic Acids Res. 2014 Feb;42(4):e22. doi: 10.1093/nar/gkt1088. Epub 2013 Nov 6. PMID: 24203706; PMCID: PMC3936733.
4、Garenne D, Thompson S, Brisson A, Khakimzhan A, Noireaux V. The all-E. coliTXTL toolbox 3.0: new capabilities of a cell-free synthetic biology platform. Synth Biol (Oxf). 2021 Aug 4;6(1):ysab017. doi: 10.1093/synbio/ysab017. PMID: 34712841; PMCID: PMC8546610.
5、Dudley QM, Cai YM, Kallam K, Debreyne H, Carrasco Lopez JA, Patron NJ. Biofoundry-assisted expression and characterization of plant proteins. Synth Biol (Oxf). 2021 Sep 11;6(1):ysab029. doi: 10.1093/synbio/ysab029. PMID: 34693026; PMCID: PMC8529701.
6、Hanko EKR, Paiva AC, Jonczyk M, Abbott M, Minton NP, Malys N. A genome-wide approach for identification and characterisation of metabolite-inducible systems. Nat Commun. 2020 Mar 5;11(1):1213. doi: 10.1038/s41467-020-14941-6. PMID: 32139676; PMCID: PMC7057948.
7、Quaglia D, Ebert MC, Mugford PF, Pelletier JN. Enzyme engineering: A synthetic biology approach for more effective library generation and automated high-throughput screening. PLoS One. 2017 Feb 8;12(2):e0171741. doi: 10.1371/journal.pone.0171741. PMID: 28178357; PMCID: PMC5298319.
8、医学分子生物学实验技术/韩骅,高国全主编.一4版.—北京:人民卫生出版社,2020.9(2021.9重印)
- 标签:贝克曼库尔特
- 收藏(0) 赞(0) 踩(0)
-
 随时了解更多仪器资讯,求购、招标、中标信息实时更新,厂商招商信息随时看。大量、齐全、专业的仪器信息尽在仪器网(yiqi.com)。扫一扫关注仪器网官方微信,随时随地查看仪器用户采购、招标需求!
随时了解更多仪器资讯,求购、招标、中标信息实时更新,厂商招商信息随时看。大量、齐全、专业的仪器信息尽在仪器网(yiqi.com)。扫一扫关注仪器网官方微信,随时随地查看仪器用户采购、招标需求!
-
为你精选
-
 铜价迭创新高导致下游需求遭到抑制 仪器行业如何度过铜资源紧缺危机?
在生产过程中,通过精益生产、自动化升级等措施提高生产效率,降低单位产品成本。同时,优化产品设计,减少不必要的铜材料使用,提高产品的市场竞争力。
2024-04-29 16:08:11 业内交流
铜价迭创新高导致下游需求遭到抑制 仪器行业如何度过铜资源紧缺危机?
在生产过程中,通过精益生产、自动化升级等措施提高生产效率,降低单位产品成本。同时,优化产品设计,减少不必要的铜材料使用,提高产品的市场竞争力。
2024-04-29 16:08:11 业内交流 -
 江苏跨境电商进出口额实现快速增长 哪些仪器有望受益跨境电商?
今年以来,江苏大力推动跨境电子商务高质量发展。 持续提升货运专线服务能力和覆盖范围,打造特色跨境电商产业带。 跨境电商进出口额实现快速增长。
2024-04-16 11:20:44 业内交流
江苏跨境电商进出口额实现快速增长 哪些仪器有望受益跨境电商?
今年以来,江苏大力推动跨境电子商务高质量发展。 持续提升货运专线服务能力和覆盖范围,打造特色跨境电商产业带。 跨境电商进出口额实现快速增长。
2024-04-16 11:20:44 业内交流 -
 华为助力金融行业加速拥抱AI 仪器行拥抱AI有哪些机遇和发展?
据华为数据存储微信ID显示,4月11日,以“数字创造未来,智慧金融”为主题的2024华为金融创新数据基础设施峰会在重庆举行。 在本次峰会上,华为数据存储产品线总裁周跃峰正式发布了金融行业四大解决方案。
2024-04-15 10:45:52 业内交流
华为助力金融行业加速拥抱AI 仪器行拥抱AI有哪些机遇和发展?
据华为数据存储微信ID显示,4月11日,以“数字创造未来,智慧金融”为主题的2024华为金融创新数据基础设施峰会在重庆举行。 在本次峰会上,华为数据存储产品线总裁周跃峰正式发布了金融行业四大解决方案。
2024-04-15 10:45:52 业内交流 -
 国家发改委确立煤炭产能储备制度 有序建设储备煤矿项目哪些仪器将受益?
我们需要了解什么是煤炭产能储备制度。这是一种政府为了应对可能出现的能源供应短缺或其他突发情况,而提前储备一定数量的煤炭生产能力的制度。这需要在正常生产之外,再额外建设一些煤矿,以备不时之需。
2024-04-12 16:58:33 业内交流
国家发改委确立煤炭产能储备制度 有序建设储备煤矿项目哪些仪器将受益?
我们需要了解什么是煤炭产能储备制度。这是一种政府为了应对可能出现的能源供应短缺或其他突发情况,而提前储备一定数量的煤炭生产能力的制度。这需要在正常生产之外,再额外建设一些煤矿,以备不时之需。
2024-04-12 16:58:33 业内交流 -
 央行加大对住房保障体系经融支持力度 地产重构经营模式带来哪些仪器行业机遇?
住房保障体系的完善和房地产市场的稳定发展,将促进相关产业链的拓展和完善。其中,检测和认证服务将成为重要的一环。从建材的质量检测到室内空气环境的评估,都需要专业的仪器设备来完成。
2024-04-07 13:30:11 业内交流
央行加大对住房保障体系经融支持力度 地产重构经营模式带来哪些仪器行业机遇?
住房保障体系的完善和房地产市场的稳定发展,将促进相关产业链的拓展和完善。其中,检测和认证服务将成为重要的一环。从建材的质量检测到室内空气环境的评估,都需要专业的仪器设备来完成。
2024-04-07 13:30:11 业内交流
-
贝克曼库尔特合成生物学自动化方案汇总

①本文由仪器网入驻的作者或注册的会员撰写并发布,观点仅代表作者本人,不代表仪器网立场。若内容侵犯到您的合法权益,请及时告诉,我们立即通知作者,并马上删除。
②凡本网注明"来源:仪器网"的所有作品,版权均属于仪器网,转载时须经本网同意,并请注明仪器网(www.yiqi.com)。
③本网转载并注明来源的作品,目的在于传递更多信息,并不代表本网赞同其观点或证实其内容的真实性,不承担此类作品侵权行为的直接责任及连带责任。其他媒体、网站或个人从本网转载时,必须保留本网注明的作品来源,并自负版权等法律责任。
④若本站内容侵犯到您的合法权益,请及时告诉,我们马上修改或删除。邮箱:hezou_yiqi
- 助力科研 成就四环冻干
- 24节气 | 人间初夏,万物并秀
- 立夏丨时至立夏,万物繁茂
- 交错人字形混合器芯片制备空间稳定的脂质体:脂质组分和PEG脂质含量的影响
- 助力石油天然气行业规范化!《岩样核磁共振参数实验室测量规范》正式发布启用
- 产品推荐 | 反射多模光纤QR系列
- 公司团建,共创美好未来;团队携手,共筑辉煌明天(日照篇)
- 【摄影大赛】五一劳动节·群星闪耀时!
- 【客户焦点】帕维亚大学探索血小板生产的生物打印之路
- NEW Webinar气候智能农业:推进作物胁迫表型研究以提高作物抗逆性
- 产品推荐 | RMS3000微型拉曼光谱仪
- 文献分享 | 快速鉴别肠炎沙门氏菌的四种血清型: 大拉曼和手持式拉曼光谱仪的性能对比
- 应用案例 | 负载型金属簇异质结构对偶氮加氢催化的表面电荷效应





